library(tidyverse)
library(ranger)
library(rsample)
library(ISLR)O objetivo deste post é apresentar aplicações relevantes das funções da família mapdo pacote purrr. Essas funções são implementadas sob o paradigma de programação funcional. Saber utilizar esse tipo de programação facilita muito o processamento de dados e modelagem. É recomendável fazer o download da cheatsheet a seguir e consultar a documentação em https://purrr.tidyverse.org/.

Além da documentação, para quem prefere acompanhar por vídeo, o Caio Lente gravou um vídeo bem detalhado sobre o assunto (para assistir, clique aqui).
Neste post serão utilizados os seguintes pacotes (o purrr já é carregado com o tidyverse):
Introdução à família map
Uma grande facilidade que se encontra no R é o fato de termos operações vetorizadas de forma nativa. Considere o exemplo a seguir, em que temos um vetor numérico e queremos saber a raíz quadrada de cada elemento. Poderíamos executar essa tarefa diretamente com sqrt.
(x <- c(1, 4, 9, 16, 25))[1] 1 4 9 16 25sqrt(x)[1] 1 2 3 4 5Note que essa função está fazendo a seguinte operação:
ou seja, aplicando a função f (raiz quadarada) a cada elemento do vetor x. De forma alternativa, podemos utilizar a função map.
map(x, sqrt)[[1]]
[1] 1
[[2]]
[1] 2
[[3]]
[1] 3
[[4]]
[1] 4
[[5]]
[1] 5No entanto, note que agora temos um objeto do tipo lista como saída e isso é importante porque a lista no R pode conter diferentes tipos de objetos. Por exemplo, não é possível ter um vetor contendo elementos inteiros, booleanos e string, mas isso é possível quando trabalhamos com uma lista. Veja no exemplo abaixo.
c(1, TRUE, "teste")[1] "1" "TRUE" "teste"list(1, TRUE, "teste")[[1]]
[1] 1
[[2]]
[1] TRUE
[[3]]
[1] "teste"Em muitas situações queremos que a saída já esteja de acordo com um tipo determinado de objeto. Por exemplo, podemos ter interesse em aplicar a função sqrt a cada elemento do objeto x com a função map, mas queremos que a saída seja diretamente um vetor do tipo double. Assim, podemos utilizar a função map_dbl.
map_dbl(x, sqrt)[1] 1 2 3 4 5Para obter os resultados em tipos específicos, podemos utilizar os seguintes pósfixos:
_dbl: vetor com elementos de números com casas decimais
_int: vetor com elementos de números inteiros
_chr: vetor com elementos de texto
_lgl: vetor com elementos booleanos (TRUE ou FALSE)
_dfc: data frame criado ao unir colunas
_dfr: data frame criado ao unir linhas
Pensando em um contexto de análise de dados, em que temos uma estrutura do tipo data frame, podemos aplicar alguma função diretamente a cada elemento do data frame. Perceba a diferença dos objetos retornados com map e map_dbl.
tibble(x = c(1, 4, 9, 16, 25)) %>%
mutate(raiz = map(x, sqrt))# A tibble: 5 × 2
x raiz
<dbl> <list>
1 1 <dbl [1]>
2 4 <dbl [1]>
3 9 <dbl [1]>
4 16 <dbl [1]>
5 25 <dbl [1]>tibble(x = c(1, 4, 9, 16, 25)) %>%
mutate(raiz = map_dbl(x, sqrt))# A tibble: 5 × 2
x raiz
<dbl> <dbl>
1 1 1
2 4 2
3 9 3
4 16 4
5 25 5Anteriormente foram apresentados funções recebendo apenas um argumento. Isso porque as funções da família map aceitam apenas um argumento como entrada. Para utilizar dois argumentos, considere a família map2.
map2 (funções que recebem dois argumentos)
Em diversos cenários precisamos trabalhar com funções com mais de um argumento. Considere um exemplo em que queremos calcular a área de diferentes retângulos a partir de um vetor com as bases e outro com as alturas. Poderíamos utilizar a função map2 da seguinte forma:
base <- c(1, 2, 3)
altura <- c(4, 5, 6)
map2(base, altura, prod)[[1]]
[1] 4
[[2]]
[1] 10
[[3]]
[1] 18A partir de uma estrutura de banco de dados, considere que queremos obter a soma das variáveis num1 e num2 para cada elemento. Aqui consideraremos a função map2 e map2_int.
tibble(num1 = 1:5,
num2 = -15:-11) %>%
mutate(soma = map2(num1, num2, sum))# A tibble: 5 × 3
num1 num2 soma
<int> <int> <list>
1 1 -15 <int [1]>
2 2 -14 <int [1]>
3 3 -13 <int [1]>
4 4 -12 <int [1]>
5 5 -11 <int [1]>tibble(num1 = 1:5,
num2 = -15:-11) %>%
mutate(soma = map2_int(num1, num2, sum))# A tibble: 5 × 3
num1 num2 soma
<int> <int> <int>
1 1 -15 -14
2 2 -14 -12
3 3 -13 -10
4 4 -12 -8
5 5 -11 -6Note que podemos fazer isso de forma diferente, mas o objetivo aqui é facilitar a compreensão e aplicação das funções da família map.
Quando a função precisa receber 3 ou mais argumentos, podemos utilizar a função pmap.
pmap (funções que recebem 3 ou mais argumentos)
Considere o cenário em que queremos obter, para cada elemento do banco de dados, a soma dos valores de três colunas (num1, num2 e num3). Podemos utilizar as funções pmap e pmap_dbl.
tibble(num1 = 1:5,
num2 = -15:-11,
num3 = seq(10, 50, 10)) %>%
mutate(soma = pmap(list(num1, num2, num3), sum))# A tibble: 5 × 4
num1 num2 num3 soma
<int> <int> <dbl> <list>
1 1 -15 10 <dbl [1]>
2 2 -14 20 <dbl [1]>
3 3 -13 30 <dbl [1]>
4 4 -12 40 <dbl [1]>
5 5 -11 50 <dbl [1]>tibble(num1 = 1:5,
num2 = -15:-11,
num3 = seq(10, 50, 10)) %>%
mutate(soma = pmap_dbl(list(num1, num2, num3), sum))# A tibble: 5 × 4
num1 num2 num3 soma
<int> <int> <dbl> <dbl>
1 1 -15 10 -4
2 2 -14 20 8
3 3 -13 30 20
4 4 -12 40 32
5 5 -11 50 44Em muitas situações, criaremos uma função para um único processamento. Para evitar a criação de um objeto desnecessário, utilizaremos a estrutura de fórmula que será convertida em uma função diretamente dentro do map. Nesse caso, temos três formas diferentes de nos referir aos argumentos:
Argumento único com . - f(x) = x + 2
tibble(numero = c(1, 4, 9, 16, 25)) %>%
mutate(raiz = map_dbl(numero, ~ .x + 2))# A tibble: 5 × 2
numero raiz
<dbl> <dbl>
1 1 3
2 4 6
3 9 11
4 16 18
5 25 27Dois argumentos com .x e .y - f(x, y) = x + y
tibble(numero = c(1, 4, 9, 16, 25),
valor = c(10, 3, 4, 5, 8)) %>%
mutate(raiz = map2_dbl(numero, valor, ~ .x + .y))# A tibble: 5 × 3
numero valor raiz
<dbl> <dbl> <dbl>
1 1 10 11
2 4 3 7
3 9 4 13
4 16 5 21
5 25 8 33Três ou mais argumentos com ..1, ..2, ..3 etc - f(x, y, z) = z (x + y)
tibble(numero = c(1, 4, 9, 16, 25),
valor = c(10, 3, 4, 5, 8),
fator = c(.1, .5, .9, .7, .2)) %>%
mutate(raiz = pmap_dbl(list(numero, valor, fator), ~ ..3 * (..1 + ..2)))# A tibble: 5 × 4
numero valor fator raiz
<dbl> <dbl> <dbl> <dbl>
1 1 10 0.1 1.1
2 4 3 0.5 3.5
3 9 4 0.9 11.7
4 16 5 0.7 14.7
5 25 8 0.2 6.6Aplicações
Nessa seção apresentaremos duas aplicações importantes da família map no contexto de machine learning e de simulação estatística. A aplicação em machine learning nos permite fazer o ajuste de hiperparâmetros para os casos em que não exista alguma implementação pronta.
Ajuste de hiperparâmetro por validação cruzada
Nessa aplicação, como base, utilizaremos o conjunto de dados Credit do pacote ISLR. O objetivo desse banco de dados é predizer os valores da variável Balance.
dados <- ISLR::Credit
head(dados)| ID | Income | Limit | Rating | Cards | Age | Education | Gender | Student | Married | Ethnicity | Balance |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 14.891 | 3606 | 283 | 2 | 34 | 11 | Male | No | Yes | Caucasian | 333 |
| 2 | 106.025 | 6645 | 483 | 3 | 82 | 15 | Female | Yes | Yes | Asian | 903 |
| 3 | 104.593 | 7075 | 514 | 4 | 71 | 11 | Male | No | No | Asian | 580 |
| 4 | 148.924 | 9504 | 681 | 3 | 36 | 11 | Female | No | No | Asian | 964 |
| 5 | 55.882 | 4897 | 357 | 2 | 68 | 16 | Male | No | Yes | Caucasian | 331 |
| 6 | 80.180 | 8047 | 569 | 4 | 77 | 10 | Male | No | No | Caucasian | 1151 |
Suponha que será utilizado um modelo de Floresta Aleatória e deseja-se definir o hiperparâmetro mtry (define o número de variáveis que serão sorteadas para cada divisão dos nós). Queremos testar os valores 2, 4, 8 e 10 para essa quantidade e, consequentemente, estimar qual desses valores leva a um menor erro de previsão. Para isso, utilizaremos a validação cruzada. A função vold_cv nos auxiliará nesse processo (veja o post Descartes e rsample para mais detalhes sobre a função vfold_cv).
set.seed(15)
vfold_cv(dados, v = 10) # v indica o número de lotes# 10-fold cross-validation
# A tibble: 10 × 2
splits id
<list> <chr>
1 <split [360/40]> Fold01
2 <split [360/40]> Fold02
3 <split [360/40]> Fold03
4 <split [360/40]> Fold04
5 <split [360/40]> Fold05
6 <split [360/40]> Fold06
7 <split [360/40]> Fold07
8 <split [360/40]> Fold08
9 <split [360/40]> Fold09
10 <split [360/40]> Fold10Para cada uma das dez linhas são definidos conjuntos de treinamento (com 360 observações) e conjuntos de teste (40 observações). O lote é identificado pela coluna id. Faremos a combinação desses lotes com os valores experimentais de mtry.
vfold_cv(dados, v = 10) %>%
crossing(mtry = c(2, 4, 8, 10))# A tibble: 40 × 3
splits id mtry
<list> <chr> <dbl>
1 <split [360/40]> Fold01 2
2 <split [360/40]> Fold01 4
3 <split [360/40]> Fold01 8
4 <split [360/40]> Fold01 10
5 <split [360/40]> Fold02 2
6 <split [360/40]> Fold02 4
7 <split [360/40]> Fold02 8
8 <split [360/40]> Fold02 10
9 <split [360/40]> Fold03 2
10 <split [360/40]> Fold03 4
# ℹ 30 more rowsAssim, para cada linha do tibble acima, ajustaremos a Floresta Aleatória com o conjunto de treino do split considerando o mtry da respectiva linha e avaliaremos o erro no conjunto de teste. Isso será feito com a aplicação de uma função para cada linha do tibble com map_dbl.
Primeiro, precisaremos definir a função para calcular o erro quadrático médio nessa situação.
ajuste <- function(splits, variaveis_mtry) {
treino <- training(splits)
teste <- testing(splits)
rf <- ranger(Balance ~ . - ID, mtry = variaveis_mtry, data = treino)
eqm <- mean((teste$Balance - predict(rf, teste)$predictions)^2)
return(eqm)
}Pronto! Agora, para obter o erro estimado em cada cenário, aplicaremos a função ajustecom auxílio da função map (execute o comando a seguir linha por linha e verifique as saídas).
set.seed(321)
vfold_cv(dados, v = 10) %>%
crossing(mtry = c(2, 4, 8, 10)) %>%
mutate(eqm = map2_dbl(splits, mtry, ajuste)) %>%
group_by(mtry) %>%
summarise(eqm_medio = mean(eqm),
desv_pad = sd(eqm)) %>%
arrange(eqm_medio)# A tibble: 4 × 3
mtry eqm_medio desv_pad
<dbl> <dbl> <dbl>
1 10 9950. 3959.
2 8 10318. 4039.
3 4 14760. 5630.
4 2 27114. 8353.Portanto, o valor de mtry que levou a menor estimativa pontual do erro quadrático médio foi de 10. Dessa forma, utilizaremos 10 variáveis em cada split das árvores da Floresta Aleatória.
E se tivéssemos como objetivo avaliar o ajuste do hiperparâmetro mtry e min.node.size? Poderíamos utilizar a função pmap_dlb da seguinte forma:
ajustes2 <- function(splits, variaveis_mtry, node_size) {
treino <- training(splits)
teste <- testing(splits)
rf <- ranger(Balance ~ . - ID, mtry = variaveis_mtry, min.node.size = node_size, data = treino)
eqm <- mean((teste$Balance - predict(rf, teste)$predictions)^2)
return(eqm)
}
set.seed(321)
vfold_cv(dados, v = 10) %>%
crossing(mtry = c(2, 4, 8, 10),
node_size = c(5, 10, 15)) %>%
mutate(eqm = pmap_dbl(list(splits, mtry, node_size), ajustes2)) %>%
group_by(mtry, node_size) %>%
summarise(eqm_medio = mean(eqm),
desv_pad = sd(eqm)) %>%
arrange(eqm_medio)# A tibble: 12 × 4
# Groups: mtry [4]
mtry node_size eqm_medio desv_pad
<dbl> <dbl> <dbl> <dbl>
1 10 5 9876. 3804.
2 8 5 10158. 3967.
3 10 10 10490. 3926.
4 8 10 11347. 4598.
5 10 15 11838. 4680.
6 8 15 12312. 5127.
7 4 5 14523. 5623.
8 4 10 15948. 6101.
9 4 15 17730. 6611.
10 2 5 26583. 8001.
11 2 10 29425. 9239.
12 2 15 32271. 9782.Logo, nesse cenário, o ideal seria utilizar mtry = 10 e node_size = 5.
Note como podemos facilitar diversas tarefas apenas com funções do pacote rsample e com a definição de funções simples.
A seguir veremos algumas aplicações para entender um pouco mais sobre a influência de certas quantidades em conceitos estatísticos.
Intervalo de Confiança
Nesta simulação trabalharemos com intervalos de confiança para a média com dados de uma distribuição normal com variância conhecida. Lembre-se que o intervalo de confiança para esse caso é dado por:
em que
Inicialmente consideraremos uma amostra (n_amostra) de tamanho 100 de uma população com média (media_pop) igual a 50, desvio padrão (dp_pop) igual a 3 e um nível de confiança (confianca) de 95%. Vamos criar a função simulacao para retornar a média amostral e os limites inferior e superior do intervalo.
media_pop <- 50
dp_pop <- 3
n_amostra <- 100
confianca <- .95
simulacao <- function(n_amostra, media_pop, dp_pop, confianca = .95) {
x <- rnorm(n_amostra, media_pop, dp_pop)
tibble(media_amostral = mean(x),
lim_inf = mean(x) - qnorm((1 + confianca)/2) * dp_pop / sqrt(n_amostra),
lim_sup = mean(x) + qnorm((1 + confianca)/2) * dp_pop / sqrt(n_amostra))
}Agora, aplicaremos a função simulacao 100 vezes. Veja a saída.
tibble(b = 1:10^2) %>%
mutate(resultado = map(b, ~simulacao(n_amostra, media_pop, dp_pop, confianca))) %>%
unnest(cols = resultado) # A tibble: 100 × 4
b media_amostral lim_inf lim_sup
<int> <dbl> <dbl> <dbl>
1 1 50.4 49.8 51.0
2 2 50.2 49.7 50.8
3 3 50.2 49.6 50.8
4 4 50.1 49.5 50.7
5 5 50.1 49.5 50.6
6 6 49.9 49.3 50.5
7 7 49.5 48.9 50.0
8 8 49.6 49.0 50.2
9 9 50.5 49.9 51.0
10 10 50.1 49.5 50.7
# ℹ 90 more rowsIdentificaremos com uma cor vermelha os intervalos que não contenham a média populacional identificada pela linha azul tracejada. Qual a porcentagem de intervalos contendo a média populacional você esperaria?
tibble(b = 1:10^2) %>%
mutate(resultado = map(b, ~simulacao(n_amostra, media_pop, dp_pop, confianca))) %>%
unnest(cols = resultado) %>%
mutate(contem = case_when(media_pop >= lim_inf & media_pop <= lim_sup ~ "contém",
TRUE ~ "não contém")) %>%
ggplot(aes(b, media_amostral, color = contem)) +
geom_hline(yintercept = media_pop, color = "blue", lty = 2) +
geom_errorbar(aes(ymin = lim_inf, ymax = lim_sup)) +
labs(x = "simulação", y = NULL, color = NULL) +
scale_color_manual(values = c("contém" = "black", "não contém" = "red")) +
theme_bw() +
theme(legend.position = "none")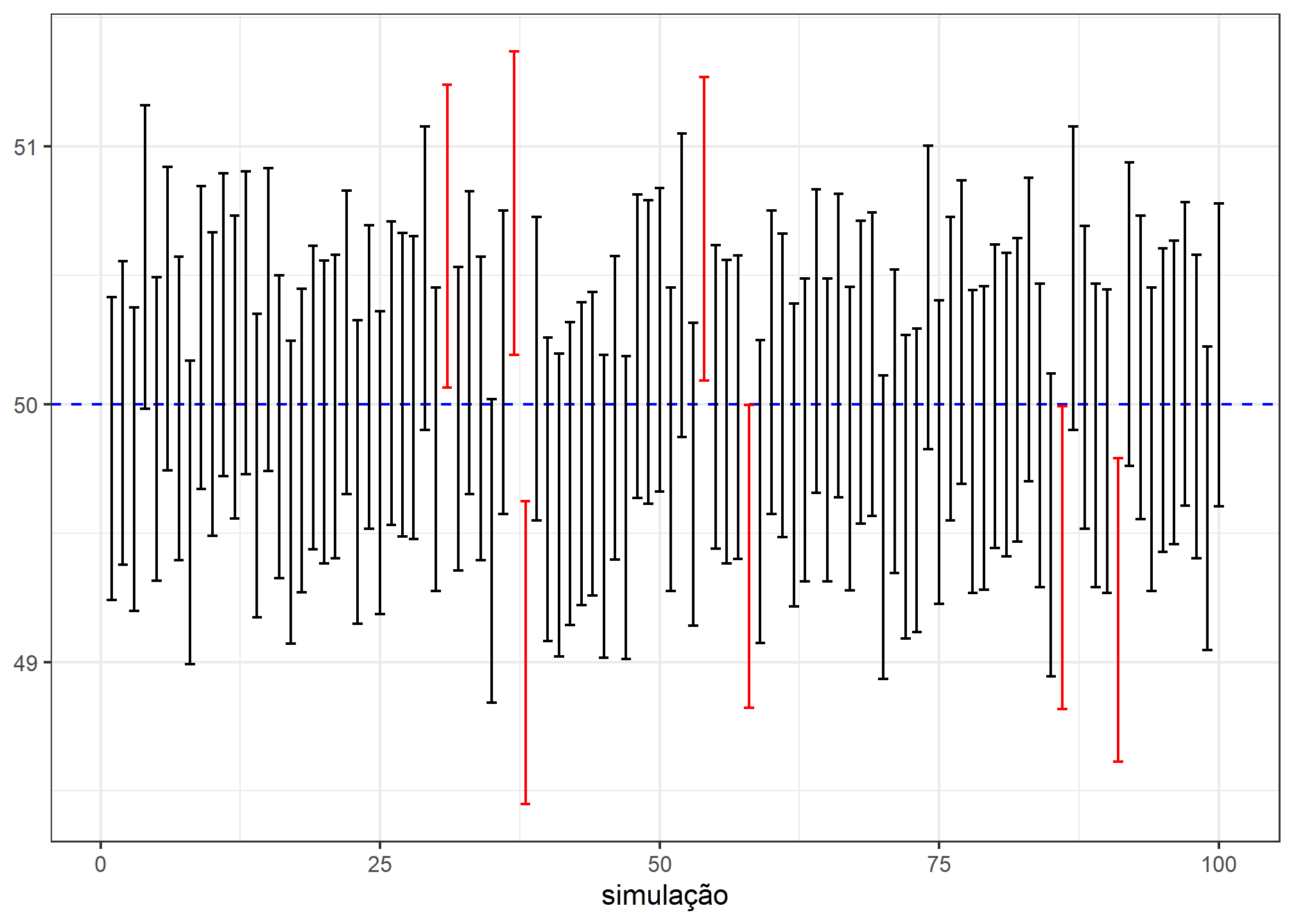
Intervalo de confiança - variando o nível de confiança
O que aconteceria com os intervalos se considerássemos diferentes níveis de confiança? Um nível de confiança maior levaria a um intervalo de amplitude maior? Para verificar essas questões, faremos uma simulação variando essa quantidade com os conceitos apresentados.
crossing(b = 1:10^2,
confianca = c(.25, .50, .75, .99)) %>%
mutate(resultado = map(confianca, ~simulacao(n_amostra, media_pop, dp_pop, .x))) %>%
unnest(cols = resultado) %>%
mutate(contem = case_when(media_pop >= lim_inf & media_pop <= lim_sup ~ "contém",
TRUE ~ "não contém")) %>%
ggplot(aes(b, media_amostral, color = contem)) +
geom_hline(yintercept = media_pop, color = "blue", lty = 2) +
geom_errorbar(aes(ymin = lim_inf, ymax = lim_sup)) +
labs(x = "simulação", y = NULL, color = NULL) +
facet_wrap(~ confianca, ncol = 2) +
scale_color_manual(values = c("contém" = "black", "não contém" = "red")) +
theme_bw() +
theme(legend.position = "none")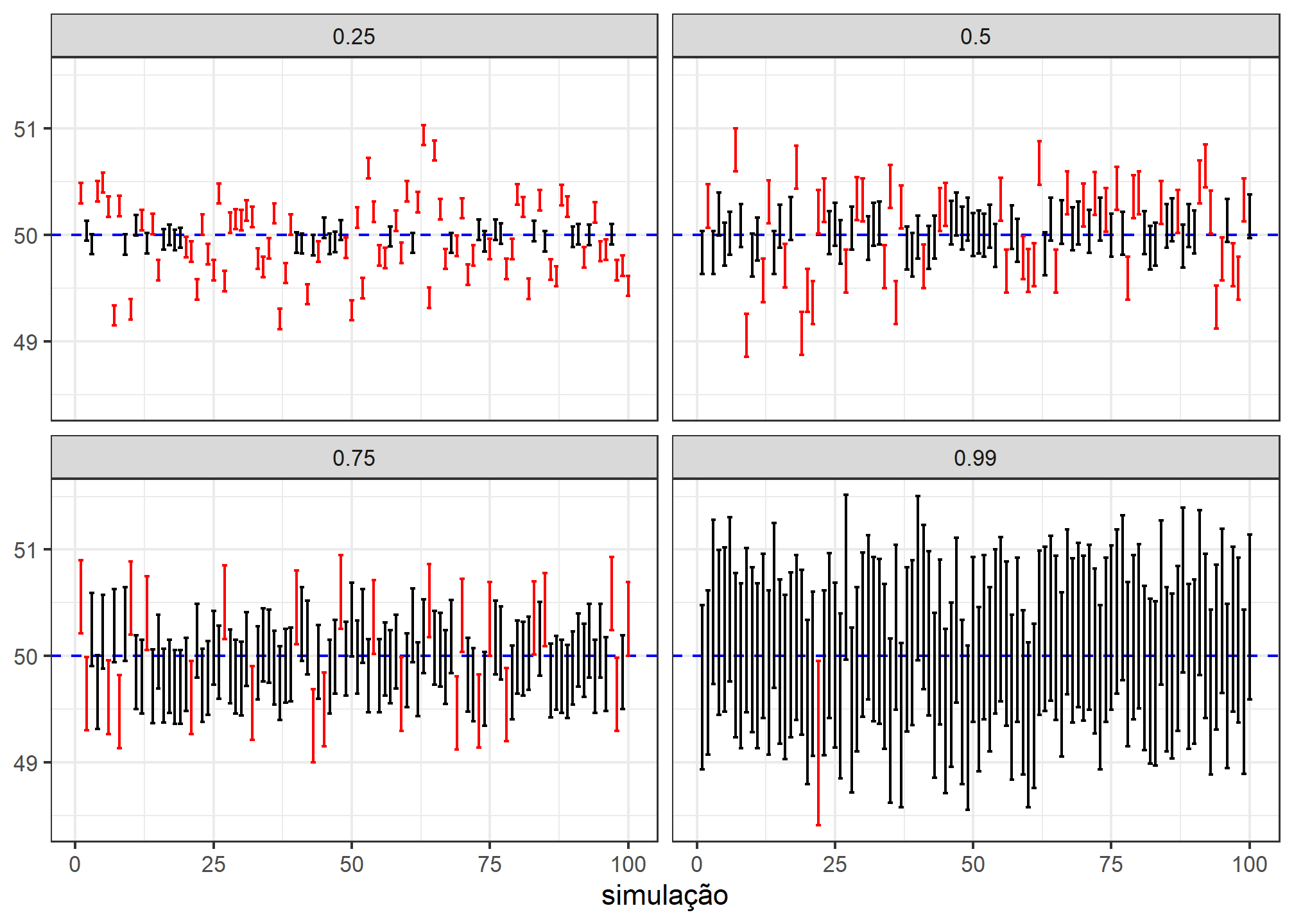
Note que baixos valores do nível de confiança estão associados a intervalos menores e um número reduzido de intervalos contendo a média populacional. Veja como os intervalos com 99% de confiança apresentam uma grande amplitude.
Intervalo de confiança - variando o tamanho amostral
E o que aconteceria com a amplitude dos intervalos ao variar o tamanho amostral fixando-se o nível de confiança?
crossing(b = 1:10^2,
n = c(10, 100, 500, 1000)) %>%
mutate(resultado = map(n, ~simulacao(.x, media_pop, dp_pop, confianca))) %>%
unnest(cols = resultado) %>%
mutate(contem = case_when(media_pop >= lim_inf & media_pop <= lim_sup ~ "contém",
TRUE ~ "não contém")) %>%
ggplot(aes(b, media_amostral, color = contem)) +
geom_hline(yintercept = media_pop, color = "blue", lty = 2) +
geom_errorbar(aes(ymin = lim_inf, ymax = lim_sup)) +
labs(x = "simulação", y = NULL, color = NULL) +
facet_wrap(~ n, ncol = 2) +
scale_color_manual(values = c("contém" = "black", "não contém" = "red")) +
theme_bw() +
theme(legend.position = "none")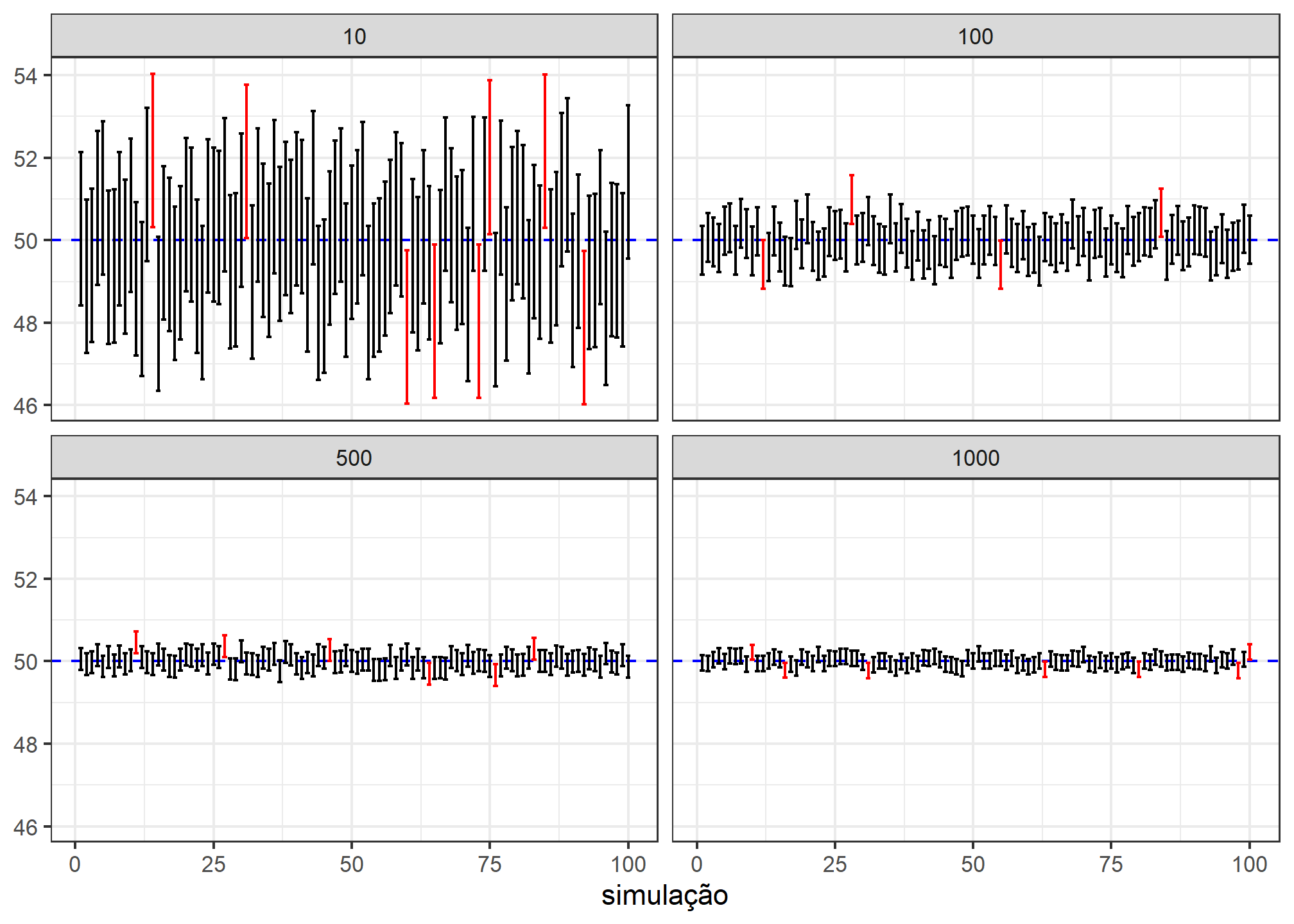
Intervalo de confiança - variando o tamanho amostral e o nível de confiança
E como se relacionam as variações do tamanho amostral e nível de confiança? A seguir apresentamos uma simulação testando diferentes combinações.
crossing(b = 1:10^2,
n = c(100, 500, 1000),
confianca = c(.33, .66, .99)) %>%
mutate(resultado = map2(n, confianca, ~simulacao(.x, media_pop, dp_pop, .y))) %>%
unnest(cols = resultado) %>%
mutate(contem = case_when(media_pop >= lim_inf & media_pop <= lim_sup ~ "contém",
TRUE ~ "não contém")) %>%
ggplot(aes(b, media_amostral, color = contem)) +
geom_hline(yintercept = media_pop, color = "blue", lty = 2) +
geom_errorbar(aes(ymin = lim_inf, ymax = lim_sup)) +
labs(x = "simulação", y = NULL, color = NULL) +
facet_wrap(n ~ confianca, ncol = 3) +
scale_color_manual(values = c("contém" = "black", "não contém" = "red")) +
theme_bw() +
theme(legend.position = "none")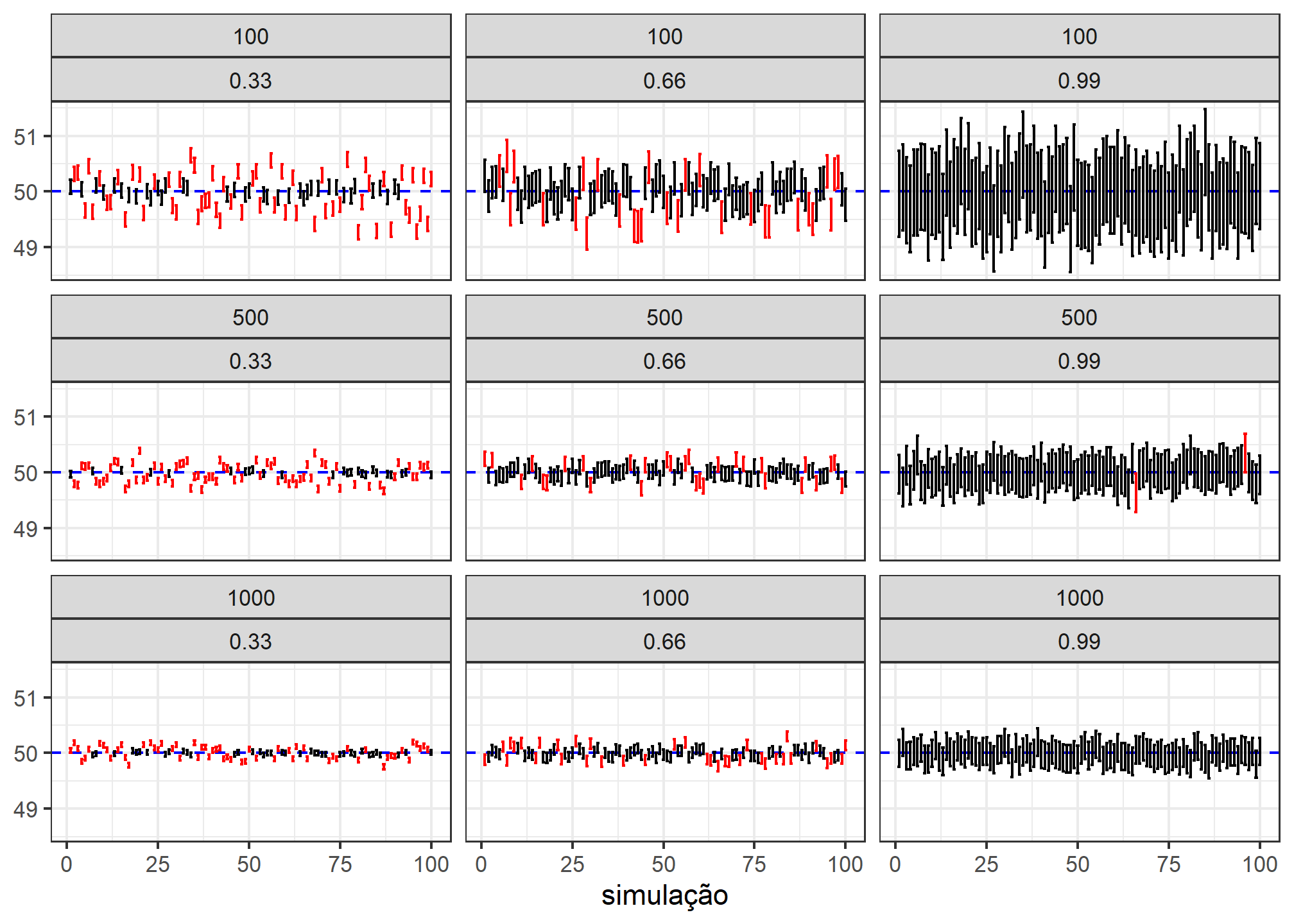
Conclusão
Nesse post aprendemos a utilizar as funções da família map com algum detalhe. Verificamos como fazer o ajuste de hiperparâmetros de forma muito rápida com auxílio do pacote rsample e como realizar diferentes simulações com poucas linhas de código. Esse último contexto de aplicação é muito útil para quem está começando a estudar estatística.
Caso tenha alguma crítica, sugestão ou comentário, me envie uma mensagem!